
Análisis de homocedasticidad en R
Index of content
Resumen:
El supuesto de homogeneidad de varianzas considera que la varianza es constante.
¿Cómo citar el presente artículo?
Romero, J. (Enero 1, 2019). Análisis de homocedasticidad en R. R.JeshuaRomeroGuadarrama. https://www.r.jeshuaromeroguadarrama.com/es/blog/applied-statistics/homoskedasticity-analysis/.
Análisis de homocedasticidad en R by Jeshua Romero Guadarrama, available under Attribution 4.0 International (CC BY 4.0) at https://www.r.jeshuaromeroguadarrama.com/es/blog/applied-statistics/homoskedasticity-analysis/.
Introducción
El supuesto de homogeneidad de varianzas, también conocido como supuesto de homocedasticidad, considera que la varianza es constante (no varía) en los diferentes niveles de un factor, es decir, entre diferentes grupos.
A la hora de realizar contrastes de hipótesis o intervalos de confianza, cuando los tamaños de cada grupo son muy distintos ocurre que:
- Si los grupos con tamaños muestrales pequeños son los que tienen mayor varianza, la probabilidad real de cometer un error de tipo I en los contrastes de hipótesis será menor de lo que se obtiene al hacer el test. En los intervalos, los límites superior e inferior reales son menores que los que se obtienen. La inferencia será por lo general más conservadora.
- Si por el contrario, son los grupos con tamaños muestrales grandes los que tienen mayor varianza, entonces se tendrá el efecto contrario y las pruebas serán más liberales. Es decir, la probabilidad real de cometer un error de tipo I es mayor que la devuelta por el test y los intervalos de confianza verdaderos serán más amplios que los calculados.
Existen diferentes test que permiten evaluar la distribución de la varianza. Todos ellos consideran como hipótesis nula que la varianza es igual entre los grupos y como hipótesis alternativa que no lo es. La diferencia entre ellos es el estadístico de centralidad que utilizan:
- Los test que trabajan con la media de la varianza son los más potentes cuando las poblaciones que se comparan se distribuyen de forma normal.
- Utilizar la media truncada mejora el test cuando los datos siguen una distribución de Cauchy (colas grandes).
- La mediana consigue mejorarlo cuando los datos siguen una distribución asimétrica.
F-test (razón de varianzas)
El F-test, también conocido como contraste de la razón de varianzas, contrasta la hipótesis nula de que dos poblaciones normales tienen la misma varianza. Es muy potente, detecta diferencias muy sutiles, pero es muy sensible a violaciones de la normalidad de las poblaciones. Por esta razón, no es un test recomendable si no se tiene mucha certeza de que las poblaciones se distribuyen de forma normal.
El F-test estudia el cociente de varianzas \(\frac{\sigma^{2}_{1}}{\sigma^{2}_{2}}\), que en caso de que sean iguales, toma el valor 1.
El estadístico F empleado sigue una distribución de F-Snedecor:
$$ F= \frac{\sigma_{2}^{2}}{\sigma_{2}^{1}} \frac{\widehat{S}^{1}}{\widehat{S}^{2}} \sim F_{n_1-1, n_2-1} $$
# Importar la libreria ggplot2
library(ggplot2)
library(dplyr)
data("iris")
iris <- filter(.data = iris, Species %in% c("versicolor", "virginica"))
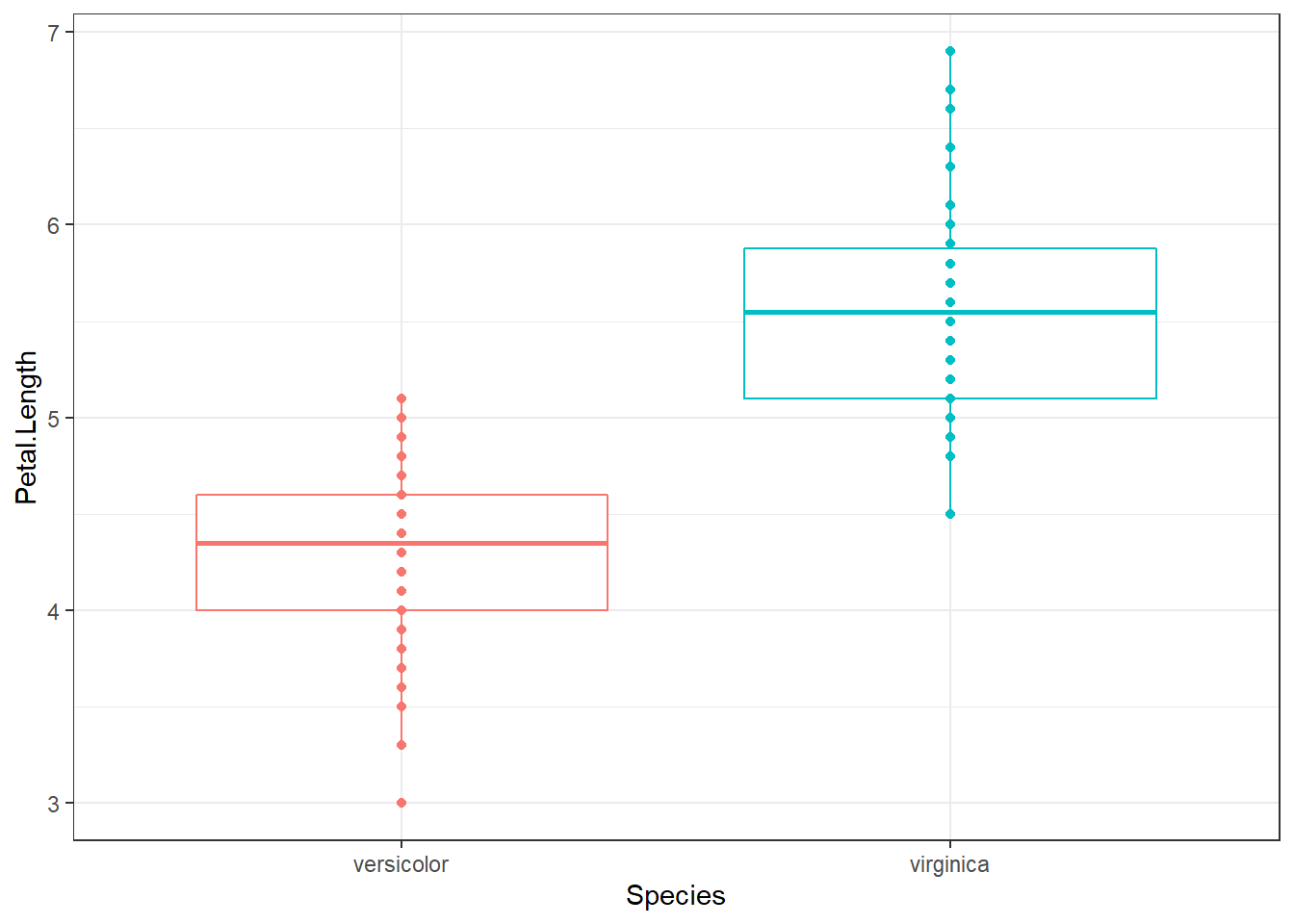
ggplot(data = iris, aes(x = Species, y = Petal.Length, colour = Species)) +
geom_boxplot() +
geom_point() +
theme_bw() +
theme(legend.position = "none")
aggregate(Petal.Length~Species, data = iris, FUN = var)
Species Petal.Length
1 versicolor 0.2208163
2 virginica 0.3045878
var.test(x = iris[iris$Species == "versicolor", "Petal.Length"],
y = iris[iris$Species == "virginica", "Petal.Length"] )
F test to compare two variances
data: iris[iris$Species == "versicolor", "Petal.Length"] and iris[iris$Species == "virginica", "Petal.Length"]
F = 0.72497, num df = 49, denom df = 49, p-value = 0.2637
alternative hypothesis: true ratio of variances is not equal to 1
95 percent confidence interval:
0.411402 1.277530
sample estimates:
ratio of variances
0.7249678
El test no encuentra diferencias significativas entre las varianzas de los dos grupos.
Test de Levene
El test de Levene se puede aplicar con la función LeveneTest() del paquete car. Se caracteriza, además de por poder comparar 2 o más poblaciones, por permitir elegir entre diferentes estadísticos de centralidad :mediana (por defecto), media, media truncada. Esto es importante a la hora de contrastar la homocedasticidad dependiendo de si los grupos se distribuyen de forma normal o no.
library(car)
data("iris")
iris <- filter(.data = iris, Species %in% c("versicolor", "virginica"))
leveneTest(y = iris$Petal.Length, group = iris$Species, center = "median")
Levene's Test for Homogeneity of Variance (center = "median")
Df F value Pr(>F)
group 1 1.0674 0.3041
98
El test no encuentra diferencias significativas entre las varianzas de los dos grupos.
Test de Bartlett
Permite contrastar la igualdad de varianza en 2 o más poblaciones sin necesidad de que el tamaño de los grupos sea el mismo. Es más sensible que el test de Levene a la falta de normalidad, pero si se está seguro de que los datos provienen de una distribución normal, es la mejor opción.
data("iris")
a <- iris[iris$Species == "versicolor", "Petal.Length"]
b <- iris[iris$Species == "virginica", "Petal.Length"]
bartlett.test(list(a,b))
Bartlett test of homogeneity of variances
data: list(a, b)
Bartlett's K-squared = 1.249, df = 1, p-value = 0.2637
El test no encuentra diferencias significativas entre las varianzas de los dos grupos.
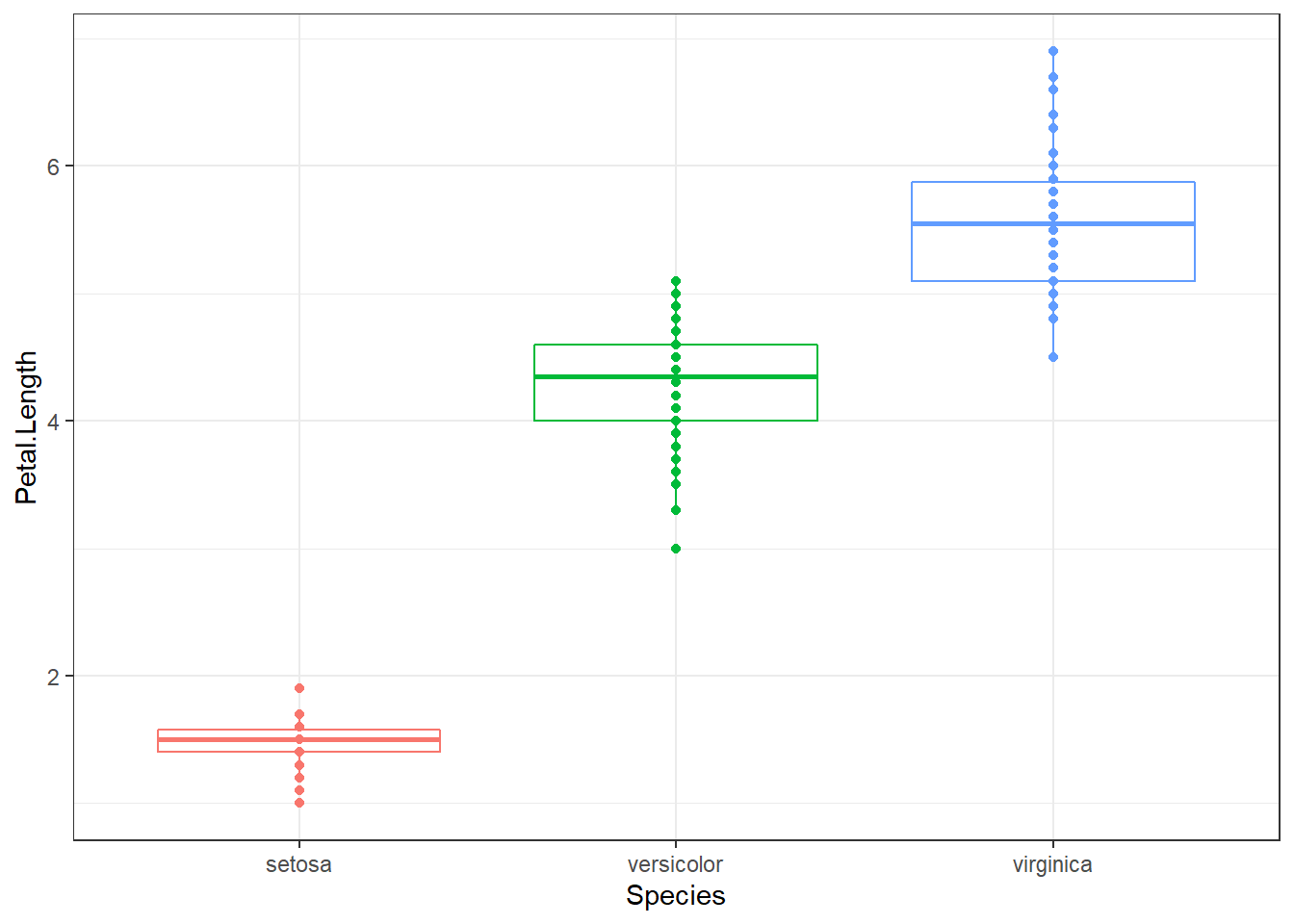
Si se aplica a los 3 grupos a la vez, sí hay evidencias de que la varianza no es la misma en todos ellos. Idea que se puede intuir a partir del tamaño de la caja y bigotes del boxplot.
data("iris")
ggplot(data = iris, aes(x = Species, y = Petal.Length, colour = Species)) +
geom_boxplot() +
geom_point() +
theme_bw() +
theme(legend.position = "none")
aggregate(Petal.Length~Species, data = iris, FUN = var)
Species Petal.Length
1 setosa 0.03015918
2 versicolor 0.22081633
3 virginica 0.30458776
bartlett.test(iris$Sepal.Length ~ iris$Species)
Bartlett test of homogeneity of variances
data: iris$Sepal.Length by iris$Species
Bartlett's K-squared = 16.006, df = 2, p-value = 0.0003345
Test de Brown-Forsyth
Se puede encontrar dentro del paquete HH, la función hov(). Es equivalente al test de Levene cuando se emplea la mediana como medida de centralidad.
# Este paquete da problemas para instalarlo en entornos linux
library(HH)
hov(iris$Sepal.Length ~ iris$Species)
hov: Brown-Forsyth
data: iris$Sepal.Length
F = 6.3527, df:iris$Species = 2, df:Residuals = 147, p-value = 0.002259
alternative hypothesis: variances are not identical
Test de Fligner-Killeen
Se trata de un test no paramétrico que compara las varianzas basándose en la mediana. Es también una alternativa cuando no se cumple la condición de normalidad en las muestras.
data("iris")
a <- iris[iris$Species == "versicolor","Petal.Length"]
b <- iris[iris$Species == "virginica","Petal.Length"]
fligner.test(x = list(a,b))
Fligner-Killeen test of homogeneity of variances
data: list(a, b)
Fligner-Killeen:med chi-squared = 1.2624, df = 1, p-value = 0.2612
Conclusión
Si se tiene seguridad de que las muestras a comparar proceden de poblaciones que siguen una distribución normal, son recomendables el F-test y el test de Bartlet, pareciendo ser el segundo más recomendable ya que el primero es muy potente pero extremadamente sensible a desviaciones de la normal.
Si no se tiene la seguridad de que las poblaciones de origen son normales, se recomiendan el test de Leven utilizando la mediana o el test no paramétrico Fligner-Killeen que también se basa en la mediana.
Referencias
- OpenIntro Statistics: Fourth Edition by David Diez, Mine Çetinkaya-Rundel, Christopher Barr libro
- Handbook of Biological Statistics by John H. McDonald
popular post

Problemas de transporte y asignación
Resumen: Existen dos tipos de problemas especiales en la investigación de operaciones, los problemas de transporte y de asignación.
Read More
Programación lineal
Resumen: Utilizar R para resolver problemas de programación lineal vinculados a la investigación de operaciones.
Read More
Programación lineal entera
Resumen: Utilizar R para resolver problemas de programación lineal entera vinculados a la investigación de operaciones.
Read More